Introducción a las bases de datos biológicas
Una de las características de la investigación genómica moderna es la generación de enormes cantidades de datos sin procesar de la secuencia. A medida que crece el volumen de los datos genómicos, sofisticadas metodologías computacionales son necesarias para administrar el diluvio de datos. Así, el primer reto de la era de la genómica es almacenar y manejar el enorme volumen de información a través de la creación y uso de bases de datos informáticos. Este capítulo presenta algunos conceptos básicos relacionados con las bases de datos, en particular, los tipos, diseños y arquitecturas de las bases de datos biológicas. Se hace hincapié en recuperar datos de las principales bases de datos biológicas, como GenBank.
- Pero, ¿QUÉ ES UNA BASE DE DATOS?
Una base de datos es un archivo computarizado que se utiliza para almacenar y organizar datos de tal manera que la información se puede recuperar fácilmente mediante una variedad de criterios de búsqueda. Las bases de datos están compuestas de hardware y software para la gestión de datos. El objetivo principal del desarrollo de una base de datos es organizar los datos en un conjunto de registros estructurados para permitir una fácil recuperación de la información. Cada registro, también denominado entrada, debe contener una serie de campos que contengan los elementos de datos reales, por ejemplo, campos para nombres, números de teléfono, direcciones, fechas, ...
Para recuperar un registro en particular de la base de datos, un usuario debe especificar una información en particular, llamada valor, que debe encontrarse en un campo en particular y esperar que la computadora recupere todo el registro de datos. Este proceso se llama hacer una consulta. Aunque el objetivo principal de todas las bases de datos es la recuperación de datos, las bases de datos biológicas a menudo tienen un mayor nivel de requisitos, conocido como descubrimiento de conocimiento, que se refiere a la identificación de conexiones entre piezas de información que no se conocían cuando se ingresó la información por primera vez. Por ejemplo, las bases de datos que contienen información de secuencia bruta pueden realizar tareas extracontrolables para identificar la homología de secuencia o los motivos conservados. Estas características facilitan el descubrimiento de nuevas perspectivas biológicas a partir de datos brutos.
- TIPOS DE BASES DE DATOS
Originalmente, todas las bases de datos utilizaban un formato de archivo plano, que es un archivo de texto largo que contiene muchas entradas separadas por un delimitador, un carácter especial como una barra vertical (|). Dentro de cada entrada hay una serie de campos separados por tabulaciones o comas. Excepto por los valores sin procesar en cada campo, el archivo de texto completo no contiene instrucciones ocultas para que las computadoras busquen información específica o creen informes basados en ciertos campos de cada registro. El archivo de texto puede considerarse una sola tabla. Por lo tanto, para buscar en un archivo plano una determinada información, una computadora debe leer todo el archivo, un proceso obviamente ineficiente. Esto es manejable para una base de datos pequeña, pero a medida que aumenta el tamaño de la base de datos o los tipos de datos se vuelven más complejos, este estilo de base de datos puede ser muy difícil para la recuperación de información. De hecho, las búsquedas en archivos electrónicos a menudo causan fallos en todo el sistema informático debido a que la naturaleza de la operación requiere mucha memoria. Para facilitar el acceso y recuperación de datos, se han desarrollado sofisticados programas informáticos para organizar, buscar y acceder a los datos. Se les llama sistemas de gestión de bases de datos. Estos sistemas no solo contienen registros de datos en bruto, sino también instrucciones operativas para ayudar a identificar conexiones ocultas entre registros de datos. El propósito de establecer una estructura de datos es facilitar la ejecución de las búsquedas y combinar diferentes registros para formar los informes finales de búsqueda. Según los tipos de estructuras de datos, estos sistemas de administración de bases de datos se pueden clasificar en dos tipos: sistemas de administración de bases de datos relacionales y sistemas de administración de bases de datos orientados a objetos. En consecuencia, las bases de datos que emplean estos sistemas de gestión se conocen como bases de datos relacionales o bases de datos orientadas a objetos, respectivamente.
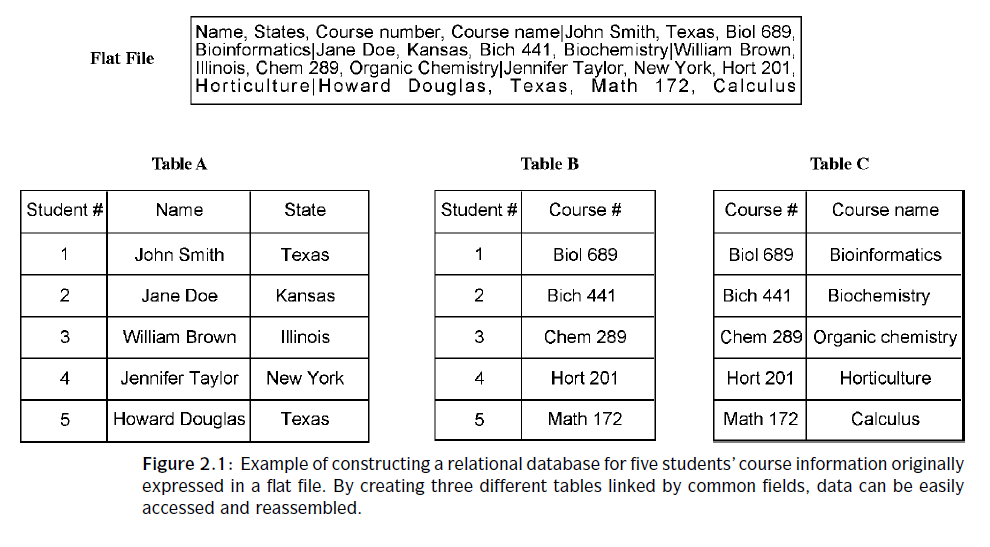
3.1. Bases de datos relacionales
En lugar de usar una sola tabla como en una base de datos de archivos planos, las bases de datos relacionales usan un conjunto de tablas para organizar los datos. Cada tabla, también llamada relación, está formada por columnas y filas. Las columnas representan campos individuales. Las filas representan valores en los campos de registros. Las columnas de una tabla están indexadas de acuerdo con una característica común llamada atributo, por lo que pueden ser referenciadas en otras tablas. Para ejecutar una consulta en una base de datos relacional, el sistema selecciona elementos de datos vinculados de diferentes tablas y combina la información en un informe. Por lo tanto, la información específica se puede encontrar más rápidamente en una base de datos relacional que en una base de datos de archivos planos. Las bases de datos relacionales se pueden crear utilizando un lenguaje de programación especial llamado lenguaje de consulta estructurado (SQL). La creación de este tipo de bases de datos puede requerir mucha planificación durante la fase de diseño. Después de la creación de la base de datos original, se puede agregar fácilmente una nueva categoría de datos sin necesidad de modificar todas las tablas existentes. La posterior búsqueda en la base de datos y la recopilación de datos para los informes son relativamente sencillas. Este es un ejemplo simple de la información del curso del estudiante expresada en un archivo plano que contiene registros de cinco estudiantes de cuatro estados diferentes, cada uno de los cuales toma un curso diferente (Fig. 2.1). Cada registro de datos, separado por una barra vertical, contiene cuatro campos que describen el nombre, el estado, el número del curso y el título. También se crea una base de datos relacional para almacenar la misma información, en la que los datos se estructuran como una serie de tablas. La Figura 2.1 muestra cómo funciona la base de datos relacional. En cada tabla, los datos que se ajustan a un criterio particular se agrupan juntos. Las diferentes tablas se pueden vincular por categorías de datos, que facilitan la búsqueda de información específica.
Por ejemplo, si uno es para hacer la pregunta, ¿qué cursos están tomando los estudiantes de Texas? La base de datos primero encontrará el campo para ''Estado'' en la Tabla A y buscará Texas. Esto devuelve a los alumnos 1 y 5. Los números de los alumnos se agrupan en la Tabla B, en la que los alumnos 1 y 5 corresponden a Biol 689 y Matemáticas 172, respectivamente. Los nombres de los cursos listados por números de cursos se encuentran en la Tabla C. Al ir a la Tabla C, se pueden recuperar los nombres de cursos exactos correspondientes a los números de los cursos. Luego se da un informe final que muestra que los tejanos están tomando los cursos de Bioinformática y Cálculo. Sin embargo, la ejecución de la misma consulta a través del archivo plano requiere que la computadora lea todo el archivo de texto palabra por palabra y almacene la información en un espacio de memoria temporal y luego marque los registros de datos que contienen la palabra Texas. Esto es fácilmente realizable para una pequeña base de datos, pero a la hora de realizar consultas en una gran base de datos utilizando archivos planos obviamente se convierte en una tarea larga y tediosa para el sistema informático.
3.2. Bases de datos orientadas a objetos
Uno de los problemas con las bases de datos relacionales es que las tablas utilizadas no describen relaciones jerárquicas complejas entre los elementos de datos. Para superar el problema, se han desarrollado bases de datos orientadas a objetos que almacenan datos como objetos. En un lenguaje de programación orientado a objetos, un objeto puede considerarse como una unidad que combina datos y rutinas matemáticas que actúan sobre los datos. La base de datos está estructurada de tal manera que los objetos están vinculados por un conjunto de punteros que definen relaciones predeterminadas entre los objetos. La búsqueda de la base de datos implica navegar a través de los objetos con la ayuda de los punteros que vinculan diferentes objetos. Los lenguajes de programación como C ++ se utilizan para crear bases de datos orientadas a objetos.
El sistema de base de datos orientado a objetos es más flexible; los datos se pueden estructurar en base a relaciones jerárquicas. Al hacerlo, las tareas de programación se pueden simplificar para los datos que se conocen para tener relaciones complejas, como los datos multimedia. Sin embargo, este tipo de sistema de base de datos carece de la base matemática rigurosa de las bases de datos relacionales. También existe el riesgo de que algunas de las relaciones entre los objetos puedan tergiversarse. Por lo tanto, algunas bases de datos actuales han incorporado características de ambos tipos de programación de bases de datos, creando el sistema de gestión de bases de datos objeto-relacional.
La información del curso de los estudiantes anterior (Fig. 2.1) se puede utilizar para construir una base de datos orientada a objetos. Se pueden diseñar tres objetos diferentes: objeto de alumno, objeto de curso y objeto de estado. Sus interrelaciones están indicadas por líneas con flechas (Fig. 2.2). Para responder a la misma pregunta, qué cursos están tomando los estudiantes de Texas, uno simplemente necesita comenzar desde Texas en el objeto estatal, que tiene indicadores que llevan a los estudiantes 1 y 5 en el objeto del estudiante. Otros indicadores en el objeto del estudiante apuntan al curso que está tomando cada uno de los dos estudiantes. Por lo tanto, una simple navegación a través de los objetos vinculados proporciona un informe final.
- BASES DE DATOS BIOLÓGICAS
Las bases de datos biológicas actuales utilizan los tres tipos de estructuras de base de datos: archivos planos, relacionales y orientados a objetos. A pesar de los inconvenientes obvios del uso de archivos planos en la administración de bases de datos, muchas bases de datos biológicas aún utilizan este formato. La justificación de esto es que este sistema implica una cantidad mínima de diseño de base de datos y los biólogos que trabajan pueden comprender fácilmente la salida de búsqueda.
Según sus contenidos, las bases de datos biológicas se pueden dividir aproximadamente en tres categorías: bases de datos primarias, bases de datos secundarias y bases de datos especializadas. Las bases de datos primarias contienen datos biológicos originales. Son archivos de secuencia bruta o datos estructurales presentados por la comunidad científica. GenBank y Protein Data Bank (PDB) son ejemplos de bases de datos primarias. Las bases de datos secundarias contienen información procesada computacionalmente o curada manualmente, basada en información original de bases de datos primarias. Las bases de datos de secuencias de proteínas traducidas que contienen anotaciones funcionales pertenecen a esta categoría. Los ejemplos son SWISS-Prot y Protein Information Resources (PIR) (sucesor del Atlas de Margarita Dayhoff de Secuencia y Estructura de Proteínas. Las bases de datos especializadas son aquellas que atienden a un interés de investigación particular. Por ejemplo, Flybase, la base de datos de secuencias de VIH y el proyecto de base de datos Ribosomal son bases de datos que se especializan en un organismo en particular o en un tipo de datos en particular. En la tabla se proporciona una lista de algunas bases de datos de uso frecuente.
4.1. Bases de datos primarias
Existen tres bases de datos de secuencias públicas principales que almacenan datos de secuencias de ácido rawnucleico producidos y enviados por investigadores de todo el mundo: GenBank, la base de datos del Laboratorio Europeo de Biología Molecular (EMBL) y el Banco de Datos de ADN de Japón (DDBJ), que están disponibles gratuitamente en Internet. La mayoría de los datos en las bases de datos son aportados directamente por autores con un mínimo nivel de anotación. Un pequeño número de secuencias, especialmente las publicadas en la década de 1980, fueron ingresadas manualmente desde la literatura publicada por el personal de administración de la base de datos. En la actualidad, el envío de secuencias a GenBank, EMBL o DDBJ es una condición previa para la publicación en la mayoría de las revistas científicas para garantizar que los datos moleculares fundamentales estén disponibles de forma gratuita. Estas tres bases de datos públicas colaboran estrechamente e intercambian nuevos datos diariamente. Juntos constituyen la colaboración internacional de bases de datos de secuencias de nucleótidos. Esto significa que, al conectarse a cualquiera de las tres bases de datos, uno debe tener acceso a los mismos datos de secuencia de nucleótidos. Aunque las tres bases de datos contienen todas las mismas series de datos en bruto, cada una de las bases de datos individuales tiene un tipo de formato ligeramente diferente para representar los datos. Afortunadamente, para las estructuras tridimensionales de las macromoléculas biológicas, solo hay una base de datos centralizada, la PDB. Esta base de datos archiva coordenadas atómicas de macromoléculas (proteínas y ácidos nucleicos) determinadas por cristalografía de rayos X y RMN. Utiliza un formato de archivo plano para representar el nombre de la proteína, los autores, los detalles experimentales, la estructura secundaria, los cofactores y las coordenadas atómicas. La interfaz web de PDB también proporciona herramientas de visualización para la manipulación simple de imágenes.
4.2. Bases de datos secundarias
La información de anotación de secuencia en la base de datos primaria a menudo es mínima. Para convertir la información de secuencia en bruto en un conocimiento biológico más sofisticado, se necesita mucho procesamiento posterior de la información de secuencia. Esto implica la necesidad de bases de datos secundarias, que contienen información de secuencia procesada computacionalmente derivada de las bases de datos primarias. La cantidad de trabajo de procesamiento computacional varía mucho entre las bases de datos secundarias; algunos son archivos simples de datos de secuencia traducidos de marcos de lectura abiertos identificados en ADN, mientras que otros proporcionan anotaciones adicionales e información relacionada con niveles más altos de información con respecto a la estructura y las funciones. Un ejemplo destacado de las bases de datos secundarias es SWISS-PROT, que proporciona anotaciones de secuencia detalladas que incluyen estructura, función y asignación de la familia de proteínas. Los datos de secuencia se derivan principalmente de TrEMBL, una base de datos de secuencias de ácido nucleico traducidas almacenadas en la base de datos de EMBL. La anotación de cada entrada está cuidadosamente curada por expertos humanos y, por lo tanto, es de buena calidad. La anotación de la proteína incluye función, estructura de dominio, sitios catalíticos, unión al cofactor, modificación postraduccional, información de la ruta metabólica, asociación de la enfermedad y similitud con otras secuencias. Gran parte de esta información se obtiene de la literatura científica y es ingresada por curadores de bases de datos. La anotación proporciona un valor agregado significativo a cada registro de secuencia original. El registro de datos también proporciona enlaces de referencias cruzadas a otros recursos en línea de interés. Otras características, como una redundancia muy baja y un alto nivel de integración con otras bases de datos primarias y secundarias, hacen que SWISS-PROT sea muy popular entre los biólogos. Un reciente esfuerzo por combinar SWISS-PROT, TrEMBL y PIR condujo a la creación de la base de datos UniProt, que tiene una cobertura más amplia que cualquiera de las tres bases de datos y al mismo tiempo mantiene la característica original de baja redundancia de SWISS-PROT. -referencias, y una alta calidad de anotación. También hay bases de datos secundarias que se relacionan con la clasificación de la familia de proteínas según funciones o estructuras. Las bases de datos de Pfam y Bloques contienen información de secuencias de proteínas alineadas, así como motivos y patrones derivados, que pueden utilizarse para la clasificación de familias de proteínas y la inferencia de funciones de proteínas. La base de datos DALI es una base de datos de estructura secundaria de proteínas que es vital para la clasificación de la estructura de las proteínas y el análisis de los hilos para identificar relaciones evolutivas distantes entre las proteínas.
4.3. Bases de datos especializadas
Las bases de datos especializadas normalmente sirven a una comunidad de investigación específica o un organismo específico. El contenido de estas bases de datos puede ser secuencias u otros tipos de información. Las secuencias en estas bases de datos pueden superponerse con una base de datos primaria, pero también pueden tener nuevos datos enviados directamente por los autores. Debido a que a menudo son curados por expertos en el campo, pueden tener organizaciones únicas y anotaciones adicionales asociadas con las secuencias. Muchas bases de datos de genoma que son taxonómicas específicas se encuentran dentro de esta categoría. Los ejemplos incluyen Flybase, WormBase, AceDB y TAIR (Tabla 2.1). Además, también hay bases de datos especializadas que contienen datos originales derivados del análisis funcional. Por ejemplo, la base de datos GenBank EST y Microarray Gene Expression Database en el European Bioinformatics Institute (EBI) son algunas de las bases de datos de expresión génica disponibles.
4.4. Interconexión entre bases de datos biológicas
Como se mencionó, las bases de datos primarias son repositorios centrales y distribuidores de información de secuencia y estructura en bruto. Son compatibles con casi todos los demás tipos de bases de datos biológicas de forma similar a Associated Press que proporciona noticias a los medios de noticias locales, que luego adaptan las noticias a sus necesidades particulares. Por lo tanto, en la comunidad biológica, existe una necesidad frecuente de que las bases de datos secundarias y especializadas se conecten a las bases de datos primarias y sigan cargando información de secuencia. Además, un usuario a menudo necesita obtener información de las bases de datos primarias y secundarias para completar una tarea porque la información en una sola base de datos a menudo es insuficiente. En lugar de permitir que los usuarios visiten varias bases de datos, es conveniente que las entradas en una base de datos tengan referencias cruzadas y se vinculen a entradas relacionadas en otras bases de datos que contengan información adicional. Todo esto crea una demanda para vincular diferentes bases de datos. La principal barrera para vincular diferentes bases de datos biológicas es la incompatibilidad de formatos. Las bases de datos biológicas actuales utilizan los tres tipos de estructuras de bases de datos: archivos planos, relacionales y orientados a objetos. Las estructuras de bases de datos heterogéneas limitan la comunicación entre bases de datos. Una solución para conectar en red las bases de datos es utilizar un lenguaje de especificación denominado Common Object Request Broker Architecture (COBRA), que permite que los programas de base de datos en diferentes ubicaciones se comuniquen en una red a través de un ''intermediario de interfaz''; sin tener que entender la estructura de la base de datos de cada uno. Funciona de una manera similar a HyperTextMarkup Language (HTML) para páginas web, etiquetando entradas de base de datos utilizando un conjunto de etiquetas comunes. Un protocolo similar llamado eXtensibleMarkup Language (XML) también ayuda a enlazar bases de datos. En este formato, cada registro biológico se divide en pequeños componentes básicos que están etiquetados con un agrupamiento jerárquico de etiquetas. Esta estructura de base de datos mejora significativamente la distribución e intercambio de anotaciones de secuencias complejas entre bases de datos. Recientemente, se ha desarrollado un protocolo especializado para el intercambio de datos bioinformáticos. Es el sistema de anotación distribuida, que permite que una computadora se ponga en contacto con varios servidores y recupere información de anotación de secuencia dispersa relacionada con una secuencia particular e integre los resultados en un solo informe combinado.
- PROBLEMAS DE LAS BASES DE DATOS BIOLÓGICAS
Uno de los problemas asociados con las bases de datos biológicas es el exceso de confianza en la información de secuencia y las anotaciones relacionadas, sin comprender la confiabilidad de la información. Lo que a menudo se ignora es el hecho de que hay muchos errores en las bases de datos de secuencias. También hay altos niveles de redundancia en las bases de datos de secuencia primaria. Las anotaciones de los genes también pueden ocasionalmente ser falsas o incompletas. Todos estos tipos de errores se pueden pasar a otras bases de datos, lo que provoca la propagación de errores. La mayoría de los errores en las secuencias de nucleótidos son causados por errores de secuenciación. Algunos de estos errores causan cambios de marco que dificultan la identificación de todo el gen o la traducción de proteínas. A veces, las secuencias de genes se contaminan con secuencias de vectores de clonación. En general, los errores son comunes para las secuencias producidas antes de la década de 1990; La calidad de la secuencia ha mejorado mucho desde entonces. Por lo tanto, se debe tener un cuidado excepcional al tratar con secuencias más fechadas. La redundancia es otro problema importante que afecta a las bases de datos primarias. Hay una tremenda duplicación de información en las bases de datos, por varias razones. Las causas de la redundancia incluyen el envío repetido de secuencias idénticas o superpuestas por parte de autores iguales o diferentes, la revisión de anotaciones, el volcado de datos de etiquetas de secuencia expresada (EST) y la administración deficiente de la base de datos que no detecta la redundancia. Esto hace que algunas bases de datos primarias sean excesivamente grandes y difíciles de manejar para la recuperación de información. Se han tomado medidas para reducir la redundancia. El Centro Nacional de Información Biotecnológica (NCBI) ahora ha creado una base de datos no redundante, llamada RefSeq, en la que se fusionan secuencias idénticas del mismo organismo y fragmentos de secuencia asociados en una sola entrada. Las secuencias de proteínas derivadas de las mismas secuencias de ADN se vinculan explícitamente como entradas relacionadas. Las variantes de secuencia del mismo organismo con diferencias muy pequeñas, que bien pueden ser causadas por errores de secuenciación, se tratan como entradas claramente relacionadas. Esta base de datos cuidadosamente curada puede considerarse una base de datos secundaria. Como se mencionó, la base de datos SWISS-PROT también tiene una redundancia mínima para las secuencias de proteínas en comparación con otras bases de datos. Otra forma de abordar el problema de la redundancia es crear bases de datos de agrupaciones de secuencias como UniGene que se unen en secuencias EST derivadas del mismo gen. El otro problema común es anotaciones erróneas. A menudo, la misma secuencia de genes se encuentra bajo diferentes nombres que dan como resultado múltiples entradas y confusión sobre los datos. O a la inversa, los genes no relacionados que llevan el mismo nombre se encuentran en las bases de datos. Para aliviar el problema de nombrar genes, es necesario reanudar los genes y las proteínas utilizando un conjunto de vocabulario común y controlado para describir un gen o una proteína. El objetivo es proporcionar un sistema de nombres coherente e inequívoco para todos los genes y proteínas. Un ejemplo destacado de tales sistemas es la ontología de genes. Algunas de las inconsistencias en la anotación podrían ser causadas por un genuino desacuerdo entre los investigadores en el campo; otros pueden resultar de la asignación imprudente de funciones de la proteína por parte de los remitentes de secuencias. También hay algunos errores que son simplemente causados por omisiones o errores al escribir. Los errores en la anotación pueden ser particularmente dañinos porque a la gran mayoría de las nuevas secuencias se les asignan funciones basadas en la similitud con las secuencias en las bases de datos que ya están anotadas. Por lo tanto, una anotación incorrecta puede transferirse fácilmente a todos los genes similares en toda la base de datos. Es posible que algunos de estos errores se puedan corregir a nivel informático estudiando los dominios de proteínas y las familias. Sin embargo, otros finalmente tienen que ser corregidos usando un trabajo experimental.
- RECUPERACIÓN DE INFORMACIÓN DE BASES DE DATOS BIOLÓGICAS
Como se mencionó, un objetivo principal en el desarrollo de bases de datos es proporcionar un acceso eficiente y amigable a los datos almacenados. Hay una serie de sistemas de recuperación de datos biológicos. Los sistemas de recuperación más populares para bases de datos biológicas son Entrez y Sequence Retrieval Systems (SRS) que brindan acceso a múltiples bases de datos para la recuperación de resultados de búsqueda integrados.
Para realizar consultas complejas en una base de datos a menudo se requiere el uso de operadores booleanos. Esto es para unir una serie de palabras clave utilizando términos lógicos como AND, OR y NO para indicar las relaciones entre las palabras clave utilizadas en una búsqueda. Y significa que el resultado de la búsqueda debe contener ambas palabras; O significa buscar resultados que contengan una palabra o ambas; NO excluye los resultados que contengan una de las palabras. Además, uno puede usar paréntesis () para definir un concepto si hay varias palabras y relaciones involucradas, de modo que la computadora sepa qué parte de la búsqueda debe ejecutar primero. Los elementos contenidos entre paréntesis se ejecutan primero. Se pueden usar citas para especificar una frase. La mayoría de los motores de búsqueda de bases de datos biológicas públicas usan alguna forma de esta lógica booleana.
Entrez
El NCBI desarrolló y mantiene Entrez, un sistema de recuperación de bases de datos biológicas. Es una puerta de enlace que permite búsquedas basadas en texto para una amplia variedad de datos, incluida información de secuencias genéticas anotadas, información estructural, así como citas y resúmenes, artículos completos y datos taxonómicos. La característica clave de Entrez es su capacidad para integrar información, que proviene de las referencias cruzadas entre las bases de datos NCBI basadas en relaciones preexistentes y lógicas entre entradas individuales. Esto es muy conveniente: los usuarios no tienen que visitar varias bases de datos ubicadas en lugares dispares. Por ejemplo, en una página de secuencia de nucleótidos, uno puede encontrar enlaces de referencias cruzadas a la secuencia de proteína traducida, a los datos de mapeo del genoma, o a la información bibliográfica relacionada de PubMed, y a las estructuras de proteínas, si están disponibles. El uso efectivo de Entrez requiere una comprensión de las características principales del motor de búsqueda. Hay varias opciones comunes a todas las bases de datos de NCBI que ayudan a limitar la búsqueda. Una opción es ''Límites'' que ayuda a restringir la búsqueda a un subconjunto de una base de datos en particular. También se puede configurar para restringir una búsqueda a una base de datos particular (por ejemplo, el campo para el autor o la fecha de publicación) o un tipo particular de datos (por ejemplo, ADN / ARN de cloroplasto). Otra opción es ''Vista previa / Índice'', que conecta diferentes búsquedas con los operadores booleanos y utiliza una serie de palabras clave conectadas lógicamente para realizar una búsqueda de noticias. La búsqueda también puede limitarse a un campo de búsqueda particular (por ejemplo, nombre del gen o número de acceso). La opción ''Historial'' proporciona un registro de las búsquedas anteriores para que el usuario pueda revisar, revisar o combinar los resultados de búsquedas anteriores. También hay un ''Portapapeles'' que almacena los resultados de búsqueda para verlos más tarde por un tiempo limitado. Para almacenar información en el Portapapeles, debe utilizarse la función ''Enviar al Portapapeles''. Una de las bases de datos a las que se puede acceder desde Entrez es una base de datos de literatura biomédica conocida como PubMed, que contiene resúmenes y, en algunos casos, el texto completo de casi 4.000 revistas. Una característica importante de PubMed es la recuperación de información basada en los títulos de los temas médicos (MeSH). El sistema MeSH consiste en una colección de más de 20,000 términos de vocabulario controlado y estandarizado que se utilizan para indexar artículos. En otras palabras, es un tesauro que ayuda a convertir palabras clave de búsqueda en términos estandarizados para describir un concepto. Al hacerlo, permite búsquedas ''inteligentes'' en las que se emplea un grupo de sinónimos aceptados para que el usuario no solo obtenga coincidencias exactas, sino también coincidencias relacionadas sobre el mismo tema que de otra manera podrían haberse perdido. Otra forma de ampliar la recuperación es mediante el uso de la opción ''Artículos relacionados''. PubMed utiliza un algoritmo de ponderación de palabras para identificar artículos relacionados con palabras similares en los títulos, resúmenes y MeSH. Al utilizar esta función, se pueden recuperar los artículos sobre el mismo tema que se perdieron en la búsqueda original. Para una búsqueda compleja, un usuario puede usar los operadores booleanos o una combinación de funciones de Límites y Vista previa / Índice para realizar búsquedas complejas. Alternativamente, las etiquetas de campo se pueden usar para mejorar la eficiencia de obtener los resultados de búsqueda. Las etiquetas son identificadores para cada campo y se colocan entre paréntesis. Por ejemplo, [AU] limita la búsqueda del nombre del autor y [JID] para el nombre de la revista. PubMed usa una lista de etiquetas para búsquedas de literatura. Los términos de búsqueda se pueden especificar mediante las etiquetas que están unidas por operadores booleanos. Algunas etiquetas de campo de PubMed que se usan con frecuencia se dan en la Tabla 2.2. Otra base de datos única a la que se puede acceder desde Entrez es Online Mendelian Inheritance in Man (OMIM), que es una base de datos basada en secuencias y de enfermedades genéticas humanas. Cada entrada en OMIM contiene información resumida sobre una enfermedad en particular, así como genes relacionados con la enfermedad. El texto contiene numerosos hipervínculos a citas bibliográficas, registros de secuencias primarias y loci cromosómicos de los genes de la enfermedad. La base de datos puede servir como un excelente punto de partida para estudiar genes relacionados con una enfermedad. El NCBI también mantiene una base de datos de taxonomía que contiene los nombres y las posiciones taxonómicas de más de 100,000 organismos con al menos una secuencia de nucleótidos o proteínas representada en la base de datos de GenBank. La base de datos de taxonomía tiene un esquema de clasificación jerárquica. El nivel de raíz es Archaea, Eubacteria y Eukaryota. La base de datos permite visualizar el árbol taxonómico de un organismo en particular. El árbol se basa en datos filogenéticos moleculares, a saber, los pequeños datos de ARN ribosomal.
GenBank
GenBank es la colección más completa de datos de secuencias de ácidos nucleicos anotados para casi todos los organismos. El contenido incluye ADN genómico, ARNm, ADNc, EST, datos de secuencia en bruto de alto rendimiento y polimorfismos de secuencia. También hay una base de datos GenPept para secuencias de proteínas, la mayoría de las cuales son traducciones conceptuales de secuencias de ADN, aunque un pequeño número de secuencias de aminoácidos se derivan utilizando técnicas de secuenciación de péptidos. Hay dos formas de buscar secuencias en GenBank. Una es usar palabras clave basadas en texto similares a una búsqueda en PubMed. El otro es usar secuencias moleculares para buscar por similitud de secuencia usando BLAST.
Formato de secuencia de GenBank
Para buscar en GenBank de manera efectiva el método basado en texto requiere una comprensión del formato de secuencia de GenBank. GenBank es una base de datos relacional. Sin embargo, la salida de búsqueda para los archivos de secuencia se produce como archivos planos para una fácil lectura. Los archivos sin formato resultantes contienen tres secciones: encabezado, características y entrada de secuencia (Ver Fig. de abajo). Hay muchos campos en las secciones Encabezado y Características. Cada campo tiene un identificador único para una fácil indexación por software de computadora. Comprender la estructura de los archivos de GenBank ayuda a diseñar estrategias de búsqueda efectivas. La sección del encabezado describe el origen de estas secuencias, la identificación del organismo y los identificadores únicos asociados con el registro. La línea superior de la sección Encabezado es el Locus, que contiene un identificador de base de datos único para una ubicación de secuencia en la base de datos (no un locus de cromosoma). El identificador va seguido por la longitud de la secuencia y el tipo de molécula (por ejemplo, ADN o ARN). A esto le sigue un código de tres letras para las divisiones de GenBank. Hay 17 divisiones en total, que se configuraron simplemente por conveniencia de almacenamiento de datos sin tener necesariamente una base científica rigurosa; por ejemplo, PLN para secuencias de plantas, hongos y algas; PRI para secuencias de primates; MAM para secuencias de mamíferos no importantes; BCT para secuencias bacterianas; y EST para secuencias EST. Junto a la división está la fecha en que el registro se hizo público (que es diferente de la fecha en que se enviaron los datos). La siguiente línea, "DEFINICIÓN", proporciona la información resumida del registro de la secuencia, incluido el nombre de la secuencia, el nombre y la taxonomía del organismo de origen, si se conoce, y si la secuencia es completa o parcial. A esto le sigue un número de acceso para la secuencia, que es un número único asignado a un fragmento de ADN cuando se envió por primera vez a GenBank y está asociado permanentemente con esa secuencia. Este es el número que debe citarse en las publicaciones. Tiene dos formatos diferentes: dos letras con cinco dígitos o una letra con seis dígitos. Para una secuencia de nucleótidos que se ha traducido en una secuencia de proteínas, se proporciona un nuevo número de acceso en forma de una cadena de caracteres alfanuméricos. Además del número de acceso, también hay un número de versión y un número de índice genético (gi). El propósito de estos números es identificar la versión actual de la secuencia. Si la anotación de la secuencia se revisa en una fecha posterior, el número de acceso sigue siendo el mismo, pero el número de versión se incrementa al igual que el número gi. Una secuencia de proteína traducida también tiene un número gi diferente de la secuencia de ADN de la que se deriva.
La siguiente línea en la sección Encabezado es el campo ''ORGANISMO'', que incluye la fuente del organismo con el nombre científico de la especie y, a veces, el tipo de tejido. Junto con el nombre científico se encuentra la información de clasificación taxonómica del organismo. Los diferentes niveles de la clasificación están vinculados a la base de datos de taxonomía del NCBI con descripciones más detalladas. A esto le sigue el campo ''REFERENCIA'', que proporciona la cita de publicación relacionada con la entrada de secuencia. La parte de REFERENCIA incluye información sobre el autor y el título de la obra publicada (o título provisional para una obra no publicada). El campo ''DIARIO'' incluye la información de la cita, así como la fecha de envío de la secuencia. La cita a menudo está vinculada al registro de PubMed para acceder a la información bibliográfica original. La última parte del encabezado es la información de contacto del remitente de la secuencia. La sección ''Características''; incluye información de anotación sobre el gen y el producto génico, así como las regiones de importancia biológica informadas en la secuencia, con identificadores y calificadores. El campo ''Fuente'' proporciona la longitud de la secuencia, el nombre científico del organismo y el número de identificación de la taxonomía. Alguna información opcional incluye la fuente del clon, el tipo de tejido y la línea celular. El campo ''gen'' es la información sobre la secuencia de codificación de nucleótidos y su nombre. Para las entradas de ADN, hay un campo ''CDS'', que es información sobre los límites de la secuencia que se puede traducir en aminoácidos. Para el ADN eucariótico, este campo también contiene información de las ubicaciones de los exones y se ingresan las secuencias de proteínas traducidas. La tercera sección del archivo plano es la secuencia que comienza con la etiqueta ''ORIGEN''. El formato de la visualización de la secuencia se puede cambiar al elegir las opciones en un menú desplegable de pantalla en la esquina superior izquierda. Para las inserciones, hay un informe BASE COUNT que incluye los números de A, G, C y T en la secuencia. Esta sección, tanto para secuencias de ADN como de proteínas, termina con dos barras diagonales (el símbolo ''//''). En las secuencias de proteínas de recuperación de ADN de GenBank, la búsqueda se puede limitar a diferentes campos de anotación, como ''organismo'', ''número de acceso'', ''autores'' y ''fecha de publicación''. Se puede utilizar una combinación de ''Límites'' y ''Vista previa'' / Index'' opciones como se describe. Alternativamente, se pueden usar una serie de calificadores de búsqueda, cada uno de los cuales define uno de los campos en un archivo GenBank. Los calificadores son similares, pero no iguales a las etiquetas de campo en PubMed. Por ejemplo, en GenBank, [GENE] representa el campo para el nombre del gen, [AUTH] para el nombre del autor y [ORGN] para el nombre del organismo. Los calificadores de GenBank utilizados frecuentemente, que deben estar en mayúsculas y entre paréntesis, se enumeran en la Tabla 2.3.
Formatos de secuencia alternativos
FASTA. Además del formato GenBank, hay muchos otros formatos de secuencia. FASTA es uno de los formatos de secuencia más simples y más populares porque contiene información de secuencia simple que es legible por muchos programas de análisis de bioinformática. Tiene una línea de definición única que comienza con un corchete de ángulo recto (>) seguido de un nombre de secuencia (Fig. 2.4). A veces, se puede proporcionar información adicional, como número de gi o comentarios, que están separados del nombre de la secuencia por un símbolo ''|''. La información adicional se considera opcional y los programas de análisis de secuencias la ignoran. La secuencia de la porcelana en los símbolos de una letra estándar comienza en la segunda línea. Cada línea de datos de secuencia está limitada a sesenta a ochenta caracteres de ancho. El inconveniente de este formato es que se pierde mucha información de anotación.
Sintaxis abstracta Notación Uno
Sintaxis abstracta Notation One (ASN.1) es un lenguaje de marcado de datos con una estructura diseñada específicamente para acceder a bases de datos relacionales. Describe las secuencias con cada elemento de información en un registro de secuencia separado por etiquetas, de modo que cada subporción del registro de secuencia se puede agregar fácilmente a las tablas relacionales y luego extraerse (Fig. 2.5). Aunque es más difícil de leer para las personas, este formato facilita que las computadoras filtren y analicen los datos. Este formato también facilita la transmisión e integración de datos entre bases de datos.
Conversión de formatos de secuencia
En el análisis de secuencia y el análisis filogenético, hay una necesidad frecuente de convertir entre los formatos de secuencia. Uno de los programas de computadora más populares para la conversión de formato de secuencia es Readseq, escrito por Don Gilbert en la Universidad de Indiana. Reconoce la mayoría de las formas y la forma y escribe un nuevo archivo en formato no analítico.
SRS (Sistema de recuperación de secuencias).
Es un sistema de arbitraje mantenido por el EBI, que es comparable al NCBI Entrez. No está tan integrado como Entrez, pero permite al usuario consultar varias bases de datos simultáneamente, otro buen ejemplo de integración de base de datos. También ofrece acceso directo a ciertas aplicaciones de análisis de secuencias, como la búsqueda de similitud de secuencias y la alineación de secuencias de Clustal (consulte el Capítulo 5). Las consultas se pueden iniciar utilizando la ''Búsqueda rápida de texto'' con un solo cuadro de consulta en el que se ingresa la información. También hay formularios de presentación más elaborados, el ''Formulario de consulta estándar'' y el ''Formulario de consulta extendida''. El formulario estándar permite utilizar cuatro criterios (campos), que están vinculados por operadores booleanos. La forma extendida permite que se usen muchos más criterios y campos diversificados. Los resultados de la búsqueda contienen la secuencia de consulta y la anotación de la secuencia, así como enlaces a la literatura, las vías metabólicas y otras bases de datos biológicas.
- RESUMEN
Las bases de datos son fundamentales para la investigación biológica, especialmente para estudios genómicos. El objetivo de una base de datos biológica es doble: recuperación de información y descubrimiento de conocimiento. Las bases de datos electrónicas se pueden construir como archivos planos, relacionales u orientados a objetos. Los archivos planos son archivos de texto simples y carecen de cualquier forma de organización para facilitar la recuperación de información por parte de las computadoras. Las bases de datos relacionales organizan los datos como tablas e información de búsqueda entre tablas con características compartidas. Las bases de datos orientadas a objetos organizan los datos como objetos y asocian los objetos según las relaciones jerárquicas. Las bases de datos biológicas abarcan los tres tipos. Según su contenido, las bases de datos biológicas se dividen en bases de datos primarias, secundarias y especializadas. Las bases de datos primarias simplemente archivan la secuencia o la información de la estructura; Las bases de datos secundarias incluyen un análisis adicional de las secuencias o estructuras. Las bases de datos especializadas atienden a un interés particular de investigación. Las bases de datos biológicas deben estar interconectadas para que las entradas en una base de datos se puedan vincular con las entradas relacionadas en otra base de datos. Las bases de datos NCBI accesibles a través de Entrez están entre las bases de datos más integradas. La recuperación efectiva de información implica el uso de operadores booleanos. Entrez tiene características adicionales fáciles de usar para ayudar a realizar búsquedas complejas. Una de estas opciones es utilizar Límites, Vista previa / Índice e Historial para reducir el espacio de búsqueda. Alternativamente, uno puede usar los calificadores de campo específicos de NCBI para realizar búsquedas. Para recuperar información de secuencia del GenBank NCBI, es necesario comprender el formato de los archivos de secuencia GenBank. También es importante tener en cuenta que los datos de secuencia en estas bases de datos son menos que perfectos. Hay errores de secuencia y anotación. Las bases de datos biológicas también están plagadas de problemas de redundancia. Existen varias soluciones para corregir la anotación y reducir la redundancia, por ejemplo, fusionar secuencias redundantes en una sola entrada o almacenar secuencias altamente redundantes en una base de datos separada.